谢正伟团队研发了一种基于基因指纹和深度学习的药效预测新系统
阅读次数: 发布日期:2021-06-29
2021年6月17日,北京大学谢正伟团队与合作者在Nature Biotechnology(影响因子36.6)在线发表了题目为“Prediction of drug efficacy from transcriptional profiles with deep learning”的研究论文,揭示了一种基于基因指纹和深度学习的药效预测系统(DLEPS,中文名灵素系统)在预测化合物治疗疾病方面的应用。
在该研究中,研究人员首先构建了一个神经网络,使用SMILES化学编码作为输入,以匹配L1000数据中测量的转录组变化,然后使用疾病相关基因指纹来反映特定疾病的“内在痕迹”,从而评估出化合物对疾病的潜在疗效。研究人员将这种方法和模型称为基于深度学习和基因指纹的药效预测系统。在预测基因表达变化部分,系统可以准确的预测新分子对转录组造成的影响,在训练集和测试集中,预测的表达谱变化和实验表达谱变化的平均相关性分别为0.90和0.74。
研究人员针对商用小分子数据库(包括FDA批准药物、天然产物、临床数据库)预测了治疗三种疾病(肥胖、高尿酸血症、非酒精性脂肪肝炎)的化合物。结果表明,对于肥胖,研究人员发现三种灵素系统预测化合物可降低小鼠的脂肪含量、诱导褐变基因(白色脂肪组织变成棕色脂肪组织)表达上调、减轻了小鼠的体重,达到了减肥的目的。对于高尿酸血症,灵素系统成功预测了天然产物紫苏烯,其可降低血清尿酸能力上优于四种阳性药物,还具有抗炎和抗纤维化的优势。对于非酒精性脂肪肝炎,研究人员发现三种MEK-ERK信号通路抑制剂可缓解非酒精性脂肪肝炎相关的肝脏病理,也是首次揭示了MEK-ERK通路在非酒精性脂肪肝炎中的重要作用。
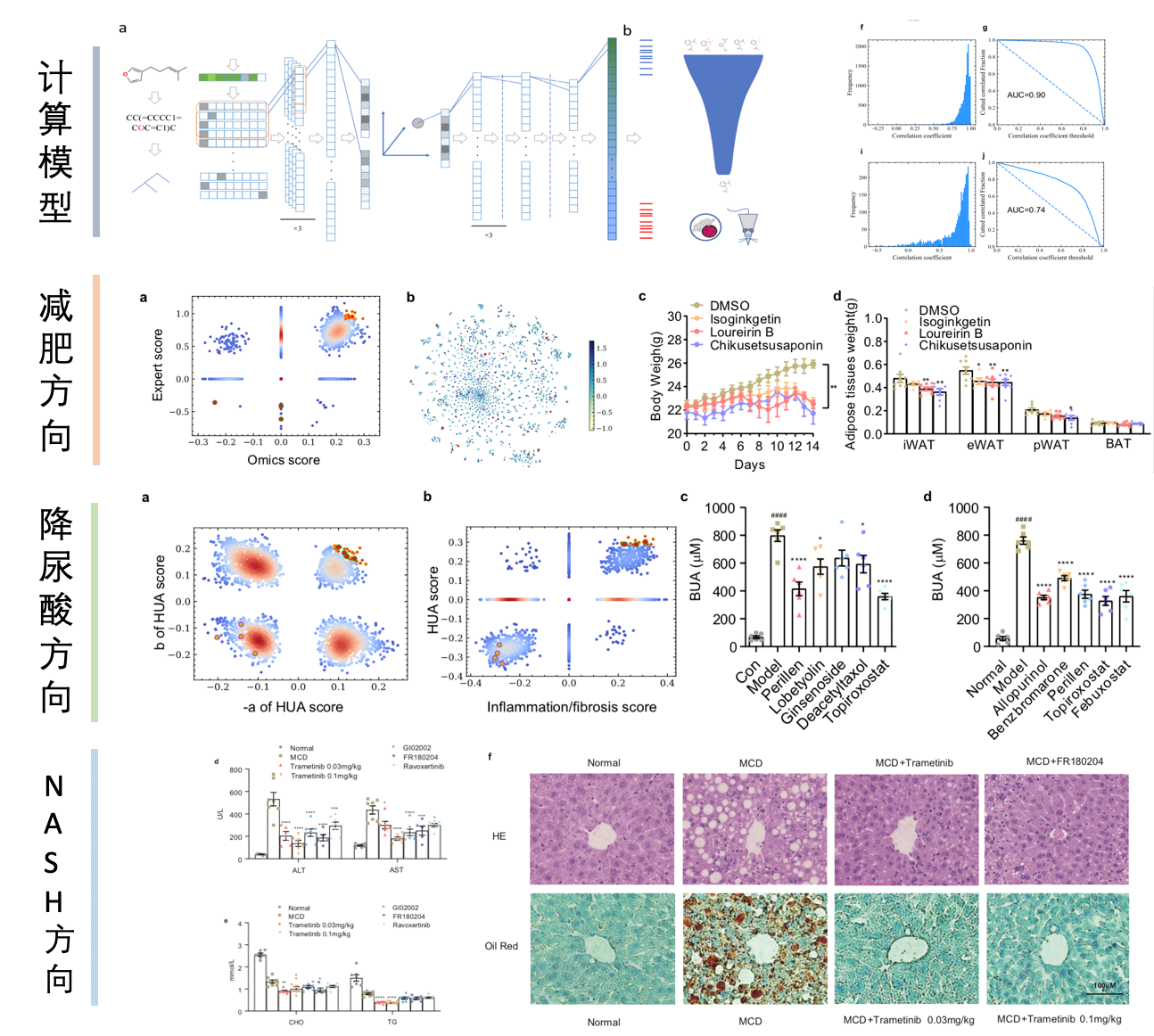
综上所述,灵素系统可成功应用于筛选治疗肥胖、高尿酸血症和NASH的化合物,总体准确率超过50%。且灵素系统可克服传统药物开发的困难,不依赖于疾病的靶点信息,仅基于基因指纹就可预测高度多样化和复杂疾病的候选分子。除在筛选疾病候选小分子方面的作用外,该研究还表明灵素系统在发现疾病致病机制以及疾病致病靶蛋白方面的潜力。随着组学的发展应用,大多数疾病,包括年龄依赖疾病、代谢紊乱和癌症,都有明确的基因标记,这为灵素系统应用于其它疾病提供了基础。值得关注的是,灵素系统对于尚无明确治疗靶点的疾病或会带来意想不到的疗效,有望成为制药行业的有利工具,为复杂疾病患者带来福音。
该研究论文投稿至Nature Biotechnology后得到了三位审稿人的高度评价。他们认为这是“一个高度原创的策略”,“是第一个用神经网络连接化合物结构和基因表达谱的报道”。论文中涉及的小分子-神经网络-基因表达变化-药效的算法框架已经申请国内外专利。
北京大学国际癌症研究院谢正伟副研究员,基础医学院郑瑞茂研究员,第一医院/国际癌症研究院张宁教授,基础医学院周虹副教授为该论文的共同通讯作者。北京大学国际癌症研究院/基础医学院硕士朱杰、郭冰冰,副主任技师王昕,亿药科技成员王靖翔、高明景为论文的共同第一作者。研究得到了国家重点研究计划合成生物学专项、国家自然科学基金、北京市自然科学基金的支持。
原文链接:https://www.nature.com/articles/s41587-021-00946-z
(北京大学国际癌症研究院)


